熱線:021-66110810,66110819
手機:13564362870


熱線:021-66110810,66110819
手機:13564362870
2.材料與方法
2.1光生物反應器的啟動和運行
為了評價在ABA系統中同時進行吡啶生物降解和脫氮的可行性,我們采用了兩個由工作體積為2.5L、高徑比為3的有機玻璃制成的管狀連續流光生物反應器(PBRs),平行運行75天,PBRs的構型如圖S1所示。LED燈安裝在PBRs周圍,以提供密度為180μmol·m-2·s-1的光,每12小時開或關一次燈。反應器運行溫度保持在25±2℃。在水力保留時間(HRT)下,將合成廢水泵入兩個PBRs。其中一個PBRs由預培養的藻-菌群接種,以PBR-0命名,而PBR-1由藻-菌群和來自城市污水處理廠(中國南京)的活性污泥接種。以副球菌(Paracoccus sp.NJUST47)和小球藻(Chlorella sorokiniana FACHB-275)為接種物,預培養48 h。副球菌為吡啶降解菌,小球藻為典型藻類。將小球藻和副球菌分別接種到PBRs中,細胞密度分別為(4.17±1.24)×106 cell·mL?1和(6.55±3.18)×106 CFU·mL?1。將活性污泥接種到PBR-1中,混合液懸浮物(MLSS)濃度為1000 mg·L?1。
合成廢水的制備方法如下:磷酸鹽緩沖液(7 mM),CaCl2·2 H2O(0.036g·L?1),MgCl2·6H2O(0.16g·L?1),碳酸鈉(0.02g·L?1)、EDTA(1mg·L?1)和微量元素溶液(1mL·L?1).磷酸二氫鉀和12水磷酸氫二鈉作為磷酸鹽緩沖液,控制pH7.2±0.1。微量元素溶液中含有硼酸(2.86g·L?1),4水氯化錳(1.81g·L?1),7水硫酸鋅(0.222g·L?1),5水硫酸銅(0.079g·L?1),NaMoO4.2H2O(0.390g·L?1),Co(NO3)2·6 H2O(0.0494g·L?1)。150mg·L?1吡啶作為唯一的有機碳和氮源加入到合成廢水中,是真實廢水中的典型濃度。相應地,吡啶和氮的負載速率為75g·m-3·d·L?1和13.3g·m-3·d·L?1,C/N比為4.3。
2.2分批式反應器和PBRs的操作
根據之前的研究,在PBR-1操作過程中進行了非原位分批實驗,研究了光照強度、額外的碳源和額外的硝酸鹽對吡啶和氮去除的影響。分批反應器由相同的200mL玻璃血清瓶建立,用帶有橡膠隔膜的鋁卷曲蓋密封,并以100rpm和25±2℃的條件放置在振動臺上。以PBR-1中提取的ABA生物量進行分批試驗接種。對于如表S1所示的分批實驗的操作條件,如果沒有特別說明的話,在合成廢水中加入濃度為150mg·L?1的吡啶,光強設置為90μmol·m-2·s-1。為了確認硝化作用在ABA體系中的作用,在50 mg·L?1烯丙基硫脲(ATU)存在下評估了吡啶和脫氮性能,該烯丙基硫脲(ATU)經常使用作為硝化抑制劑。為了驗證反硝化作用,以吡啶/硝酸鹽摩爾比為0.5的硝酸鹽作為電子受體加入。為了研究光密度的影響,間歇式反應器使用LED照明裝置在90、180和270μmol·m-2·s-1的光強度下運行。為了研究外部碳源的影響,根據完全反硝化,以1.9 mM的化學計量法加入醋酸鹽,C/N比為6.0。
2.3顯微分析
為了進行熒光原位雜交(FISH)分析,在第75天從PBR-1中提取ABA樣品,用薄刀片將ABA切割成橫截面。此后,根據Lukumbuzya等人的方法制備樣品。使用以下探針:由TAMRA標記的NSO190,對氨氧化β變形菌特異性;Ntspa662用VIC標記,靶向所有硝基螺旋菌;PAR1457由太平洋藍標記,用于識別反硝化菌;和覆蓋大多數細菌的Cy5標記的EUB338。藻類可以通過葉綠素的熒光來觀察。使用共聚焦激光掃描顯微鏡(CLSM)(A1 HD25-TI2-U,Nikon,Japan)觀察熒光。此外,使用微電極自動化系統(PA2000,Unisense,Denmark)測量光相期間ABA內的DO和pH分布。
2.4元轉錄組學和酶活性分析
在第75天從PBR-1和PBR0中提取藻類-細菌污泥樣本,以進行元轉錄組學和酶活性分析。元轉錄組測序、組裝和分析在中國馬約比奧-上海進行。簡單地說,使用E。Z.N.A.®土壤RNAMidi試劑盒(OmegaBio-tek,諾克羅斯,GA,US.),根據制造商的協議。用NanoDrop2000(賽默飛雪科學公司,US.).根據制造商的說明,使用核糖體零磁性試劑盒對樣品的總RNA進行rRNA去除程序。使用TruSeq?RNA樣本制備試劑盒(Illumina)構建cDNA文庫。根據制造商的說明,在有限公司Majorbio Bio-Pharm技術有限公司的Illumina Hiseq 2500平臺上,使用Hiseq 4000 PE Cluster Kit和Hiseq 4000SBS Kit對條形碼庫進行配對末端測序。每個樣本的開放閱讀框(ORFs)都使用轉基因掃描進行預測。所有利用CD-HIT技術將序列一致性為95%(覆蓋率為90%)的序列聚類為非冗余基因目錄。將質量控制后的Reads定位到具有95%同源性的代表性基因上,并使用RSEM評估FPKM。采用BLASTP進行非冗余基因注釋,將非冗余基因目錄與NCBINR數據庫進行對齊,e值截止值為1e?5.使用BLASTP對KEGG數據庫進行KEGG注釋。此外,通過在UniProtKB上搜索輔助基因調控因子、群體感應(QS)和自誘導器等關鍵詞,建立了本地QS數據庫。
氨單加氧酶(AMO)和羥胺氧化酶(HAO)這兩種關鍵硝化酶,以及硝酸還原酶(NAR)、亞硝酸鹽還原酶(NIR)、一氧化氮還原酶(NOR)和一氧化二氮還原酶(N2OR)這四種反硝化酶的活性,使用相應的硝化和反硝化酶聯免疫測定(ELISA)試劑盒(上海景康生物工程有限公司,中國)進行分析。
2.5分析方法
在分析前,所有的水樣在采樣后立即通過0.22μm的膜進行過濾。采用高效液相色譜法(HPLC,e2695,Waters)對吡啶進行了鑒定和定量。溶解有機碳(DOC)濃度用TOC分析儀(VARIOTOC,元素,德國)測量。NH4+-N是采用奈斯勒試劑比色法進行分析。分別采用N-(1-萘)鹽酸乙二胺分光光度法和2-異丙基-5-甲基酚分光光度法測定NO2--N和NO3--N。采用Hach試劑的過硫酸鹽氧化分光光度法測定TN。采用HPLC/MS技術鑒定吡啶生物降解中間體,并配備ABSCIEXTrapleTOF5600+HPLC系統、離子阱質譜儀和電噴霧電離源。生成的氣體混合物通過氣相色譜與FID和TCD探測器進行氣相色譜(GC-2014C,日本)進行分析。總氮(TN)包括液體中的氮(TNLiquor)和生物量中的氮(TNBio)。硝化-反硝化對TN的去除可以根據TNinitial和TNfinal之間的差異來評估。液體中的氮(TNLiquor)的去除可歸因于同化和硝化反硝化對TN的去除。同化法對TN的去除可以根據液體中的氮(TNLiquor)去除和硝化反硝化法對TN去除的差異來計算。根據進水TNLiquor(吡啶-N)和出水TNLiquor(吡啶氮、NH4+-N、NO2--N和NO3--N的總和)的差異計算TNLiquor的去除率總N計算清液量基于進水的差異TNLivul(吡啶氮)和廢水TNLivul(吡啶氮、NH4+-N、NO2--N和NO3--N的總和)。
硝化反硝化對TNLiquor的去除貢獻可以通過以下方程進行評估:
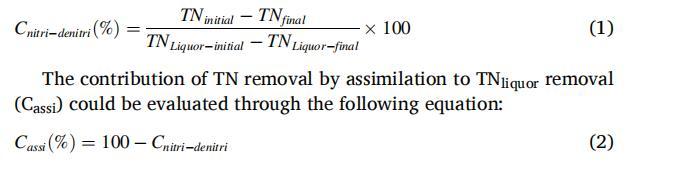
通過同化去除TN對TNLiquor去除的貢獻可以通過以下方程進行評估:
 相關新聞
相關新聞